「pythonで遺伝的アルゴリズム(GA)を実装して巡回セールスマン問題(TSP)をとく」の交叉(部分的交叉)のアルゴリズムをgolangで書き直してみた
今、GAのアルゴリズムを使った動的ルーティングのライブラリ化を試みているのですが、Geneに複雑な(というか、面倒くさい)メカニズムを組み込む為に、四苦八苦しています。
これまで私は、「順序交叉」という手法を用いてきたのですが、昨夜(の深夜)この方式では、私のやりことができないことが判明し、現在、一から作り直しています(2ヶ月分がふっとんだ感じがします)。
で、こちらのpythonで遺伝的アルゴリズム(GA)を実装して巡回セールスマン問題(TSP)をとくのページの「実装例: partial_crossover」の部分を参考させて頂いて、golangで表現してみました。
package main
import (
"fmt"
"math/rand"
)
func main() {
//var sliceA = []int{-1, 1, -1, 2, 6, 3, -1, 4, -1, 5}
//var sliceB = []int{1, -1, 2, 3, -1, 4, -1, 5, 6, -1}
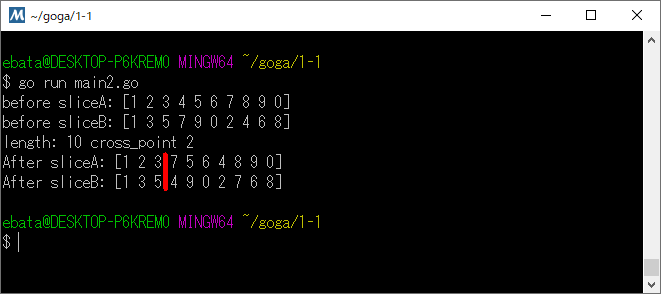
var sliceA = []int{1, 2, 3, 4, 5, 6, 7, 8, 9, 0}
var sliceB = []int{1, 3, 5, 7, 9, 0, 2, 4, 6, 8}
fmt.Println("before sliceA:", sliceA)
fmt.Println("before sliceB:", sliceB)
sliceA, sliceB = partial_crossover(sliceA, sliceB)
fmt.Println("After sliceA:", sliceA)
fmt.Println("After sliceB:", sliceB)
}
// 交叉:2つの遺伝子をランダムな位置で交叉させる
// def partial_crossover(parent1, parent2):
func partial_crossover(sliceA []int, sliceB []int) ([]int, []int) {
//num = len(parent1)
length := len(sliceA)
//cross_point = random.randrange(1, num-1)
cross_point := rand.Intn(length-2) + 1
//cross_point = 3
fmt.Println("length:", length, "cross_point", cross_point)
//child1 = parent1
sliceA1 := make([]int, length)
copy(sliceA1, sliceA)
//child2 = parent2
sliceB1 := make([]int, length)
copy(sliceB1, sliceB)
//for i in range(num - cross_point):
for i := 0; i < length-cross_point; i++ {
//target_index = cross_point + i
target_index := cross_point + 1
//target_value1 = parent1[target_index]
//target_value2 = parent2[target_index]
target_value1 := sliceA[target_index]
target_value2 := sliceB[target_index]
//exchange_index1 = np.where(parent1 == target_value2)
//exchange_index2 = np.where(parent2 == target_value1)
var exchange_index1, exchange_index2 int
for k := 0; k < length-cross_point; k++ {
if target_value2 == sliceA[k] {
exchange_index1 = k
break
}
}
for k := 0; k < length-cross_point; k++ {
if target_value1 == sliceB[k] {
exchange_index2 = k
break
}
}
//child1[target_index] = target_value2
//child2[target_index] = target_value1
//child1[exchange_index1] = target_value1
//child2[exchange_index2] = target_value2
sliceA1[target_index] = target_value2
sliceB1[target_index] = target_value1
sliceA1[exchange_index1] = target_value1
sliceB1[exchange_index2] = target_value2
}
return sliceA1, sliceB1
}
このコーディングで正解なのか分かりません(私が、バグを仕込んでいる可能性大)。
また検証した結果、私の考えている遺伝子配列(染色体の中に、遺伝子"*"を使う)では、使えないことが分かりました。
この方式では重複する遺伝子は使えないからです(というか、"*"を使う遺伝子配列は、普通ではない)。
もし利用を予定されている方は、十分に検証されることをお勧めします。
以上