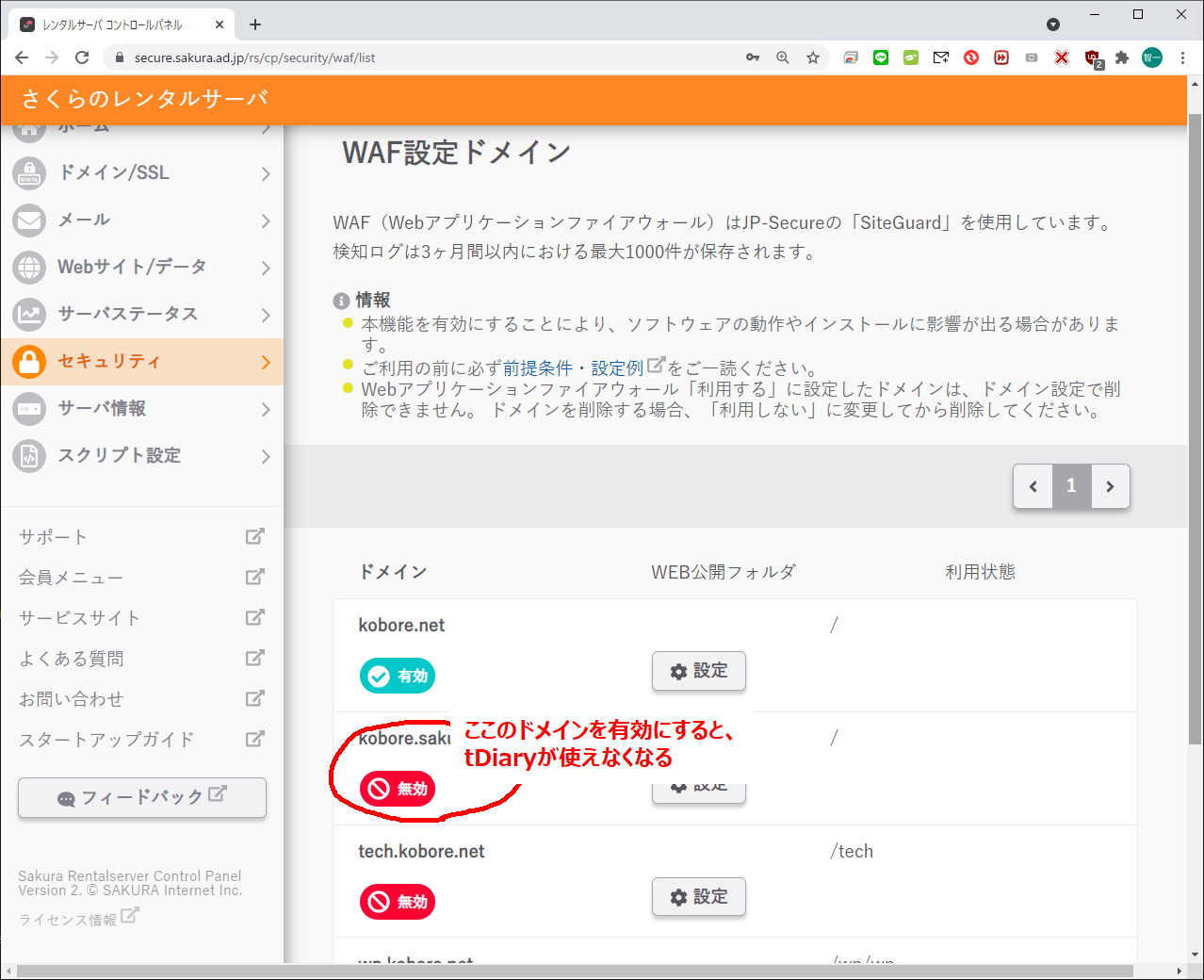
Keyword: tDiary, さくらインターネット、WAF 設定
江端智一のホームページ

Keyword: tDiary, さくらインターネット、WAF 設定
私の日記をご覧頂いている"ガジェット好きのY"さんから、メールを頂きました。
非常に有用な(特に、プライベートに使える高速計算機を欲し続けている私にとっては)情報であると思いましたので、ご本人のご許諾を頂いて、メールの(ほぼ)全文を公開させて頂きます。
江端様
日記やitmediaで執筆なされた記事をいつも楽しく拝見して
都内の中小企業で情シスとして働いております、"ガジェット好きのY"と申しま
江端様の2021年6月13日の日記を読み、まだご検討されてな
「ゲーミングマシン」を検討されていたようなので、複雑な構造力
構造力学系の計算を行うソフトウェアがGPUではなく、CPUが
ノートPCに適合する場合が多いのですが、GPUを外付け出来る
『eGPU』とは、External Graphics Processing Unitの略で、外付けのグラフィックプロセッサーのこと
- WindowsとMacの両方で利用可能。
- 接続インターフェースはThunderbolt 3。
イメージといたしましては、ノートPCに小型デスクトップぐらい
<参考URL>
https://www2.razer.com/jp-jp/g
自宅にeGPUを置いて、外では軽快なノートPCとして利用、自
金に糸目をつけないのであれば、大学にもeGPUを置いて、家と
ご検討の余地があれば、私がゲーム等で利用しているノートPCと
お伝えできる構成といたしましては、あくまでもゲームに効くGP
もちろん私に確認せずとも、ネットで検索いただければ大体の金額
お子様がご利用になられるとのことですので、上記構成等に拒絶反
以上、どうぞよろしくお願いいたします。
=========
情報を御提供頂き、誠にありがとうございます。「GPUを外付け出来るeGPU」につきましては、次女の方での
NVIDIAのGPUというキーワードから、以前購入した「CU
私は、数万から数百万のオブジェクトを使った交通シミュレーショ
http://www.iaiai.org/journals/
(正直、会社がリモートで提供するGPUなんぞ、申請の手間暇を
> ご検討の余地があれば、私がゲーム等で利用しているノートPCと
是非お願い致します。購入や使用実績があるデータは、とても役に
テキストベースで、品名(品番)と価格をお知らせ頂ければ幸いと
お手数をおかけいたしますが、何卒よろしくお願い申し上げます。
江端智一
江端様
ご返信ありがとうございます。
まず先に、私のeGPU込みのゲーム環境をお伝えいたします。
<eGPUと周辺>
1) eGPU本体:Razer Core X 39,359円(現在 40,088円)
https://www.biccamera.com/bc/i
2) GPU:玄人志向 GK-RTX3080Ti-E12GB/WHITE 213,400円(現在 237,110円)
https://www.biccamera.com/bc/i
※添付画像は1)に2)を取り付け、カバーを外した状態となりま
3) ケーブル:Thunderbolt 4 ケーブル 5,999円(現在 5,999円)
https://www.amazon.co.jp/gp/pr
<ノートPC>
4) ノートPC本体:ONE-NETBOOK ONEGX1PJR-B10 195,800円(現在 195,800円)
https://www.yodobashi.com/prod
<番外編>
5) 4Kモニター 現在はノートPC→eGPU→4kモニターとつないで、3Dゲー
6) MacBookPro 過去はMacBookPro→eGPU(中のGPUはAMD製)
構成と購入店はほとんど上記のとおりですが、2)だけ、売り切れ
GPU(グラボ)はコロナによる生産数低下からくる半導体不足と
私は正直高いと思いましたが、今買わないと手に入るのが遠のくと
CUDAプログラミングであればNVIDIA製のGPUのみとな
とは言うものの、揃えたら安くない金額で、想定した動作をしない
<ご参考>
上位モデル
RTX3090(CUDAコア数10,496) 現在 295,000円(価格.com調べ最安値)(20200924
RTX3080ti(CUDAコア数10,240) 現在
205,700円(価格.com調べ最安値)(20210603
RTX3080(CUDAコア数8,704) 現在 179,299円(価格.com調べ最安値)(20200917
RTX3070ti(CUDAコア数6,144) 現在 99,800円(価格.com調べ最安値)(20210610発
RTX3070(CUDAコア数5,888) 現在 118,800円(価格.com調べ最安値)(20201029
RTX3060ti(CUDAコア数4,864) 現在 185,332円(価格.com調べ一軒のみ)(2020120
RTX3060(CUDAコア数3,584) 現在 79,200円(価格.com調べ最安値)(20210225当
RTX2060(CUDAコア数1,920) 現在 57,800円(価格.com調べ一軒のみ)
GTX1660SUPER(CUDAコア数1,408) 現在 55,479円(価格.com調べ最安値)
下位モデル
発売当初に3080、3070、3060tiあたりを買えた人が
今後はマイニングに対するハッシュレート制限版の各モデルが販売
<ご参考2>
eGPUとPCをつなぐThunderbolt3について
デスクトップPCでThunderbolt3ポートがついている
また、追加できる拡張ボードもほぼありません。(特定メーカー専
ThunderboltポートがついているノートPCは豊富にあ
USB-C3.2等と同じコネクタ形状ですが、USB-C3.2
USB3系はダメですがUSB4はeGPUと接続可能です。(私
Thunderbolt4も搭載機種が発売され始めており、おそ
eGPUの制約(?)で短いケーブルじゃないと伝送速度が保てな
カンで上位の(Thunderbolt4)ケーブルで使えると考
GPUを直接挿入できるデスクトップPCと、ノートPC+eGP
"ガジェット好きのY"様
詳細なスペックの御説明をありがとうございました。大変参考になりました。
少なくとも、今後次女がノートPCを変更する場合には、(eGP
私の方は、これから、デスクトップにGPUを外付けする検討を開
私は、ゲームが苦手で(簡単に負けるから)ですが、GPUをシミ
可能かどうか分かりませんが、Dockerのコンテナにつっこん
<ご参考2>
Thunderbolt接続ではないeGPUについて
こちらは私も情報収集中で、まだ実施する段階にはございませんが
最近デスクトップPCにも搭載されている「M.2」スロットに、
この場合は、気軽に抜き差し出来ませんので、外付けのお手軽感は
Youtubeで「M.2 EGPU」と検索すると多数の参考動画がヒットすると思われます
いつかやってみたいなと思っております。
ひさびさにラズパイにログインして、こんなメッセージが表示されていたら、
Welcome to Ubuntu 16.04.7 LTS (GNU/Linux 4.4.38-v7+ armv7l)
* Documentation: https://help.ubuntu.com
* Management: https://landscape.canonical.com
* Support: https://ubuntu.com/advantage999 updates can be applied immediately.
To see these additional updates run: apt list --upgradableNew release '18.04.5 LTS' available.
Run 'do-release-upgrade' to upgrade to it.
『ふむ、じゃあ、アップグレードしてみようかね』って思うじゃないですか?
で、私から6時間も作業時間を奪った挙句に、
The upgrade has aborted. The upgrade needs a total of 2,827 M free space on disk '/'. Please free at least an additional 39.8 M of disk space on '/'. Empty your trash and remove temporary packages of former installations using 'sudo apt-get clean'. The upgrade has aborted. The upgrade needs a total of 49.5 M free space on disk '/boot'. Please free at least an additional 4,309 k of disk space on '/boot'. You can remove old kernels using 'sudo apt autoremove' and you could also set COMPRESS=xz in /etc/initramfs-tools/initramfs.conf to reduce the size of your initramfs.
って、メッセージを出すのは、ちょっと酷くない? って思うんですよね。
(一応、上記の指示は、全部やってみたよ、勿論)
『そんな潤沢なリソース持っとらんわ!』と捨て台詞を吐いて、今週末を終えました。
(注)以下の情報は、「轢断のシバタ」先生から御提供頂いた情報です。
■SARS-CoV-2(武漢由来のオリジナルの新型コロナ)の全ゲノム情報です(https://www.ncbi.nlm.nih.gov/nuccore/1798174254)
■スパイクタンパク質の形です(https://numon.pdbj.org/mom/246?l=ja)
■こちらはSARS-CoV-2のゲノムの解説です。この中のGene Sがスパイクタンパク、もしくはサーフェイスタンパクと呼ばれる物です。(https://plaza.umin.ac.jp/~OIO/blog/2020/05/23/full-genome-sequence-of-sars-cov-2/)
■下記が、S protein(spike protein, surface protein)のDNA配列とされる全長です。
atgtttgtttttcttgttttattgccactagtctctagtcagtgtgttaatcttacaaccagaactcaattaccccctgcatacactaattctttcacacgtggtgtttattaccctgacaaagttttcagatcctcagttttacattcaactcaggacttgttcttacctttcttttccaatgttacttggttccatgctatacatgtctctgggaccaatggtactaagaggtttgataaccctgtcctaccatttaatgatggtgtttattttgcttccactgagaagtctaacataataagaggctggatttttggtactactttagattcgaagacccagtccctacttattgttaataacgctactaatgttgttattaaagtctgtgaatttcaattttgtaatgatccatttttgggtgtttattaccacaaaaacaacaaaagttggatggaaagtgagttcagagtttattctagtgcgaataattgcacttttgaatatgtctctcagccttttcttatggaccttgaaggaaaacagggtaatttcaaaaatcttagggaatttgtgtttaagaatattgatggttattttaaaatatattctaagcacacgcctattaatttagtgcgtgatctccctcagggtttttcggctttagaaccattggtagatttgccaataggtattaacatcactaggtttcaaactttacttgctttacatagaagttatttgactcctggtgattcttcttcaggttggacagctggtgctgcagcttattatgtgggttatcttcaacctaggacttttctattaaaatataatgaaaatggaaccattacagatgctgtagactgtgcacttgaccctctctcagaaacaaagtgtacgttgaaatccttcactgtagaaaaaggaatctatcaaacttctaactttagagtccaaccaacagaatctattgttagatttcctaatattacaaacttgtgcccttttggtgaagtttttaacgccaccagatttgcatctgtttatgcttggaacaggaagagaatcagcaactgtgttgctgattattctgtcctatataattccgcatcattttccacttttaagtgttatggagtgtctcctactaaattaaatgatctctgctttactaatgtctatgcagattcatttgtaattagaggtgatgaagtcagacaaatcgctccagggcaaactggaaagattgctgattataattataaattaccagatgattttacaggctgcgttatagcttggaattctaacaatcttgattctaaggttggtggtaattataattacctgtatagattgtttaggaagtctaatctcaaaccttttgagagagatatttcaactgaaatctatcaggccggtagcacaccttgtaatggtgttgaaggttttaattgttactttcctttacaatcatatggtttccaacccactaatggtgttggttaccaaccatacagagtagtagtactttcttttgaacttctacatgcaccagcaactgtttgtggacctaaaaagtctactaatttggttaaaaacaaatgtgtcaatttcaacttcaatggtttaacaggcacaggtgttcttactgagtctaacaaaaagtttctgcctttccaacaatttggcagagacattgctgacactactgatgctgtccgtgatccacagacacttgagattcttgacattacaccatgttcttttggtggtgtcagtgttataacaccaggaacaaatacttctaaccaggttgctgttctttatcaggatgttaactgcacagaagtccctgttgctattcatgcagatcaacttactcctacttggcgtgtttattctacaggttctaatgtttttcaaacacgtgcaggctgtttaataggggctgaacatgtcaacaactcatatgagtgtgacatacccattggtgcaggtatatgcgctagttatcagactcagactaattctcctcggcgggcacgtagtgtagctagtcaatccatcattgcctacactatgtcacttggtgcagaaaattcagttgcttactctaataactctattgccatacccacaaattttactattagtgttaccacagaaattctaccagtgtctatgaccaagacatcagtagattgtacaatgtacatttgtggtgattcaactgaatgcagcaatcttttgttgcaatatggcagtttttgtacacaattaaaccgtgctttaactggaatagctgttgaacaagacaaaaacacccaagaagtttttgcacaagtcaaacaaatttacaaaacaccaccaattaaagattttggtggttttaatttttcacaaatattaccagatccatcaaaaccaagcaagaggtcatttattgaagatctacttttcaacaaagtgacacttgcagatgctggcttcatcaaacaatatggtgattgccttggtgatattgctgctagagacctcatttgtgcacaaaagtttaacggccttactgttttgccacctttgctcacagatgaaatgattgctcaatacacttctgcactgttagcgggtacaatcacttctggttggacctttggtgcaggtgctgcattacaaataccatttgctatgcaaatggcttataggtttaatggtattggagttacacagaatgttctctatgagaaccaaaaattgattgccaaccaatttaatagtgctattggcaaaattcaagactcactttcttccacagcaagtgcacttggaaaacttcaagatgtggtcaaccaaaatgcacaagctttaaacacgcttgttaaacaacttagctccaattttggtgcaatttcaagtgttttaaatgatatcctttcacgtcttgacaaagttgaggctgaagtgcaaattgataggttgatcacaggcagacttcaaagtttgcagacatatgtgactcaacaattaattagagctgcagaaatcagagcttctgctaatcttgctgctactaaaatgtcagagtgtgtacttggacaatcaaaaagagttgatttttgtggaaagggctatcatcttatgtccttccctcagtcagcacctcatggtgtagtcttcttgcatgtgacttatgtccctgcacaagaaaagaacttcacaactgctcctgccatttgtcatgatggaaaagcacactttcctcgtgaaggtgtctttgtttcaaatggcacacactggtttgtaacacaaaggaatttttatgaaccacaaatcattactacagacaacacatttgtgtctggtaactgtgatgttgtaataggaattgtcaacaacacagtttatgatcctttgcaacctgaattagactcattcaaggaggagttagataaatattttaagaatcatacatcaccagatgttgatttaggtgacatctctggcattaatgcttcagttgtaaacattcaaaaagaaattgaccgcctcaatgaggttgccaagaatttaaatgaatctctcatcgatctccaagaacttggaaagtatgagcagtatataaaatggccatggtacatttggctaggttttatagctggcttgattgccatagtaatggtgacaattatgctttgctgtatgaccagttgctgtagttgtctcaagggctgttgttcttgtggatcctgctgcaaatttgatgaagacgactctgagccagtgctcaaaggagtcaaattacattacacataa
上記のDNA配列をアミノ酸配列に翻訳すると下記のMFVFから始まる配列が得られます。それ用のソフトウェアや専用のホームページなど一瞬で変換されます。
この配列から立体構造を専門のソフトでシミュレーションした結果に得られた前述の画像などが、皆さんがニュースで見たことのあるスパイクタンパク質の絵です。
皆さんの身体にmRNAワクチンまたはウイルスベクターワクチンが投与されると、体内で下記のアミノ酸配列(=SARS-CoV-2のスパイクタンパク)の全部or一部が合成されます。
これに免疫系が反応して抗体が作られることになります。
MFVFLVLLPLVSSQCVNLTTRTQLPPAYTNSFTRGVYYPDKVFRSSVLHSTQDLFLPFFSNVTWFHAIHVSGTNGTKRFDNPVLPFNDGVYFASTEKSNIIRGWIFGTTLDSKTQSLLIVNNATNVVIKVCEFQFCNDPFLGVYYHKNNKSWMESEFRVYSSANNCTFEYVSQPFLMDLEGKQGNFKNLREFVFKNIDGYFKIYSKHTPINLVRDLPQGFSALEPLVDLPIGINITRFQTLLALHRSYLTPGDSSSGWTAGAAAYYVGYLQPRTFLLKYNENGTITDAVDCALDPLSETKCTLKSFTVEKGIYQTSNFRVQPTESIVRFPNITNLCPFGEVFNATRFASVYAWNRKRISNCVADYSVLYNSASFSTFKCYGVSPTKLNDLCFTNVYADSFVIRGDEVRQIAPGQTGKIADYNYKLPDDFTGCVIAWNSNNLDSKVGGNYNYLYRLFRKSNLKPFERDISTEIYQAGSTPCNGVEGFNCYFPLQSYGFQPTNGVGYQPYRVVVLSFELLHAPATVCGPKKSTNLVKNKCVNFNFNGLTGTGVLTESNKKFLPFQQFGRDIADTTDAVRDPQTLEILDITPCSFGGVSVITPGTNTSNQVAVLYQDVNCTEVPVAIHADQLTPTWRVYSTGSNVFQTRAGCLIGAEHVNNSYECDIPIGAGICASYQTQTNSPRRARSVASQSIIAYTMSLGAENSVAYSNNSIAIPTNFTISVTTEILPVSMTKTSVDCTMYICGDSTECSNLLLQYGSFCTQLNRALTGIAVEQDKNTQEVFAQVKQIYKTPPIKDFGGFNFSQILPDPSKPSKRSFIEDLLFNKVTLADAGFIKQYGDCLGDIAARDLICAQKFNGLTVLPPLLTDEMIAQYTSALLAGTITSGWTFGAGAALQIPFAMQMAYRFNGIGVTQNVLYENQKLIANQFNSAIGKIQDSLSSTASALGKLQDVVNQNAQALNTLVKQLSSNFGAISSVLNDILSRLDKVEAEVQIDRLITGRLQSLQTYVTQQLIRAAEIRASANLAATKMSECVLGQSKRVDFCGKGYHLMSFPQSAPHGVVFLHVTYVPAQEKNFTTAPAICHDGKAHFPREGVFVSNGTHWFVTQRNFYEPQIITTDNTFVSGNCDVVIGIVNNTVYDPLQPELDSFKEELDKYFKNHTSPDVDLGDISGINASVVNIQKEIDRLNEVAKNLNESLIDLQELGKYEQYIKWPWYIWLGFIAGLIAIVMVTIMLCCMTSCCSCLKGCCSCGSCCKFDEDDSEPVLKGVKLHYT
http://mstc.or.jp/faop/committee/11b4d681d4059ef7fed08409b8b34e1f2c1c4029.PDF
--[[ Wireshark用 NX/Dlinkプロトコル解析プログラム
ver0.03 2012年2月16日
ver0.02 2012年2月10日
ver0.01 2012年2月1日
江端智一
使用条件
絶対的な意味において無保証
History
Ver 0.03 重複登録を一応回避
Ver 0.02 TCP/UDPデータに"NUXM"があったら、送信ポート番号を調べて
動的にDissectorをWiresharkに登録するようにした
Ver 0.01 とりあえず動作確認のみ
udpポート番号が特定の値に固定されている
フィールドの位置がズレている可能性あり
使い方
(Step.1) c:/Program Files/Wireshark/init.lua の一部を書き換え
(a) disable_lua = true; do return end;
↓
-- disable_lua = true; do return end;
(b) run_user_scripts_when_superuser = false
↓
run_user_scripts_when_superuser = true
(c) 最終行に 以下の一行を追加
"init.lua"と同じディレクトリに置く
dofile("nxdlink.lua")
(Step.2) このファイルを c:/Program Files/Wireshark/ に
"nxdlink.lua"という名前で保存
]]
do
nxdlink_proto = Proto("NXDlink", "nxdlink protocol dissector")
nxdlink_proto.dissector = function(buffer, pinfo, tree)
-- nexus header type : "NUXM"
local hd_h_type_range = buffer(0,4)
local hd_h_type = hd_h_type_range:string()
-- message length ( 16K + 64 )
local hd_ml_range = buffer(4,4)
local hd_ml = hd_ml_range:uint()
-- source protocol address
local hd_sa_range = buffer(8,4)
local hd_sa = hd_sa_range:uint()
-- destination address
local hd_da_range = buffer(12,4)
local hd_da = hd_da_range:uint()
-- boot time stamp
local hd_v_seq_range = buffer(16,4)
local hd_v_seq = hd_v_seq_range:uint()
-- message number
local hd_seq_range = buffer(20,4)
local hd_seq = hd_seq_range:uint()
--[[ /* message control type */
/* UDP_MSG : multicast send */
/* UDP_INQ : multicast inquire */
/* UDP_NIQ : multicast Ninquire */
/* TCP_MSG : peer send */
/* TCP_INQ : peer inquire */
/* TCP_RPL : peer reply */]]
local hd_m_ctl_range = buffer(24,4)
local hd_m_ctl = hd_m_ctl_range:uint()
local control_type = {
[0x80000000] = "UDP_MSG(0x80000000) : multicast send",
[0xa0000000] = "UDP_INQ(0xa0000000) : multicast inquire",
[0x88000000] = "UDP_NIQ(0x88000000) : multicast Ninquire",
[0x40000000] = "TCP_MSG(0x40000000) : peer send",
[0x60000000] = "TCP_INQ(0x60000000) : peer inquire",
[0x50000000] = "TCP_RPL(0x50000000) : peer reply",
}
-- /* inquire ID parameter */
-- /* inquire source address */
local inq_id_range = buffer(28,12)
-- /* transaction code */
local hd_tcd_range = buffer(40,2)
local hd_tcd = hd_tcd_range:uint()
-- /* program version number */
local hd_ver_range = buffer(42,2)
local hd_ver = hd_ver_range:uint()
-- /* future use */
local hd_fu0_range = buffer(44,3)
local hd_fu0 = hd_fu0_range:uint()
-- /* acknowledge request mode */
-- /* PT_REQ : request message */
-- /* PT_ACK : response message */
local hd_pkind_range = buffer(47,1)
local hd_pkind = hd_pkind_range:uint()
-- /* packet seqence number */
local hd_pseq_range = buffer(48,4)
local hd_pseq = hd_pseq_range:uint()
-- /* message mode */
-- /* HEAD_ONLINE : online mode */
-- /* HEAD_TEST : test mode */
local hd_mode_range = buffer(52,2)
local hd_mode = hd_mode_range:uint()
-- /* protocol version number */
-- /* NEXUS_DLINK : NeXUS/Dlink */
-- /* NEXUS_T : NeXUS/T */
local hd_pver_range = buffer(54,1)
local hd_pver = hd_pver_range:uint()
-- /* message service level */
local hd_pri_range = buffer(55,1)
local hd_pri = hd_pri_range:uint()
-- /* current block number */
local hd_cbn_range = buffer(56,1)
local hd_cbn = hd_cbn_range:uint()
-- /* total block number */
local hd_tbn_range = buffer(57,1)
local hd_tbn = hd_tbn_range:uint()
-- /* segmenting block size */
local hd_bsize_range = buffer(58,2)
local hd_bsize = hd_bsize_range:uint()
-- /* future use */
local hd_fu1_range = buffer(60,4)
local hd_fu1 = hd_fu1_range:uint()
-- data
local data_range = buffer(64)
local data = data_range:string()
local subtree = tree:add("NX Dlink Protocol")
-- nexus header type : "NUXM"
subtree:add(hd_h_type_range, "Type:",hd_h_type)
-- message length ( 16K + 64 )
subtree:add(hd_ml_range, "Length:",hd_ml)
-- source protocol address
dispatch_addr("source protocol address:",hd_sa_range, pinfo, subtree)
-- /* destination address */
dispatch_addr("destination address:",hd_da_range, pinfo, subtree)
-- /* boot time stamp */
subtree:add(hd_v_seq_range, "boot time stamp:",hd_v_seq)
-- /* message number */
subtree:add(hd_seq_range,"message number:",hd_seq)
-- /* message control type */
dispatch_cnttype(string.format("message control type: %s",control_type[hd_m_ctl]), hd_m_ctl_range, pinfo, subtree)
-- /* inquire ID parameter */
dispatch_inq("inquire ID parameter:",inq_id_range, pinfo, subtree)
-- /* transaction code */
subtree:add(hd_tcd_range, "transaction code:",hd_tcd)
-- /* program version number */
subtree:add(hd_ver_range, "program version number:",hd_ver)
-- /* future use */
subtree:add(hd_fu0_range, "future use:",hd_fu0)
-- /* acknowledge request mode */
subtree:add(hd_pkind_range, "acknowledge request mode:",hd_pkind)
-- /* packet seqence number */
subtree:add(hd_pseq_range, "packet seqence number:",hd_pseq)
-- /* message mode */
subtree:add(hd_mode_range, "message mode(1:online 0:test) :",hd_mode)
-- /* protocol version number */
subtree:add(hd_pver_range, "NX protocol version number:",hd_pver)
-- /* message service level */
subtree:add(hd_pri_range, "message service level:",hd_pri)
-- /* current block number */
subtree:add(hd_cbn_range, "current block number:",hd_cbn)
-- /* total block number */
subtree:add(hd_tbn_range, "total block number:",hd_tbn)
-- /* segmenting block size */
subtree:add(hd_bsize_range, "segmenting block size:",hd_bsize)
-- /* future use */
subtree:add(hd_fu1_range, "future use:",hd_fu1)
-- data
subtree:add(data_range, "data:",data)
-- dispatch_inq("Data:",data_range, pinfo, subtree)
pinfo.cols.protocol = "NX/Dlink"
pinfo.cols.info = control_type[hd_m_ctl]
end
-- リスナーを定義
function init_listener()
u = {}
t = {}
u_cnt = 1;
t_cnt = 1;
u_bool = true
t_bool = true
-- UDP/TCPデータの中に"NUXM"があったらフックする
tap = Listener.new("frame", "udp contains NUXM or tcp contains NUXM")
function tap.reset()
print("passed tap.reset")
u_cnt = 0;
t_cnt = 0;
end
-- Dissector を Wireshark に追加登録
-- 重複登録問題を(不細工だけけど)以下で対応
function tap.packet(pinfo,tvb,ip)
-- UDPの場合
if ( pinfo.ipproto == 17 ) then
u_flag = 1
for i=0, u_cnt do
if u[i] == pinfo.dst_port then
u_flag = 0
end
end
if u_flag == 1 then
u_cnt = u_cnt + 1
u[u_cnt] = pinfo.dst_port
udp_table = DissectorTable.get("udp.port")
udp_table:add(pinfo.dst_port, nxdlink_proto)
end
-- TCPの場合 (まだ実験前)
elseif ( pinfo.ipproto == 6 ) then
t_flag = 1
for i=0, t_cnt do
if u[i] == pinfo.dst_port then
c_flag = 0
end
end
if t_flag == 1 then
t_cnt = t_cnt + 1
t[t_cnt] = pinfo.dst_port
tcp_table = DissectorTable.get("tcp.port")
tcp_table:add(pinfo.dst_port, nxdlink_proto)
end
end
end
end
init_listener()
end
function dispatch_cnttype(string, buffer, pinfo, subtree)
local subsubtree = subtree:add(buffer(0), string, buffer(0):tvb())
subsubtree:add(buffer(0,1),string.format("%d... .... .... .... .... .... .... .... = multicast flag",buffer(0,1):bitfield(0)))
subsubtree:add(buffer(0,1),string.format(".%d.. .... .... .... .... .... .... .... = unicast flag",buffer(0,1):bitfield(1)))
subsubtree:add(buffer(0,1),string.format("..%d. .... .... .... .... .... .... .... = inquire flag",buffer(0,1):bitfield(2)))
subsubtree:add(buffer(0,1),string.format("...%d .... .... .... .... .... .... .... = reply flag",buffer(0,1):bitfield(3)))
subsubtree:add(buffer(3,1),string.format(".... .... .... .... .... .... .... .%d.. = ack flag(future use)",buffer(3,1):bitfield(5)))
subsubtree:add(buffer(3,1),string.format(".... .... .... .... .... .... .... ...%d = ack flag(future use)",buffer(3,1):bitfield(7)))
end
function dispatch_udp_port(string, buffer, pinfo, subtree)
local subsubtree = subtree:add(buffer(0), string, buffer(0):tvb())
subsubtree:add(buffer(0,2),"source port:", buffer(0,2):uint())
subsubtree:add(buffer(2,2),"destination port:", buffer(2,2):uint())
end
function dispatch_addr(string, buffer, pinfo, subtree)
local subsubtree = subtree:add(buffer(0), string, buffer(0):tvb())
subsubtree:add(buffer(0,1),"Domain Number:", buffer(0,1):uint())
subsubtree:add(buffer(1,1),"Data Field Number:", buffer(1,1):uint())
subsubtree:add(buffer(2,2),"Node Number/Multicast Group Number:", buffer(2,2):uint())
end
function dispatch_inq(string, buffer, pinfo, subtree)
local subsubtree = subtree:add(buffer(0), string, buffer(0):tvb())
subsubtree:add(buffer(0,2), "inquire source address:",buffer(0,2):uint())
subsubtree:add(buffer(2,2), "inquire control block address:",buffer(2,2):uint())
subsubtree:add(buffer(4,2), "inquire ID sequence number:",buffer(4,2):uint())
end
「Open Street Map: マップ上にマーカー表示と経度・緯度の取得するには」のコードを使わせて頂いて、OpenStreetMap API 等の勉強をさせて頂いています。
今日は、マウスクリックで、始点と終点の位置情報を取れる方法を試してみました。
<!DOCTYPE html>
<html lang="ja">
<head>
<meta charset="utf-8">
<title>Open Street Map Test</title>
<style type="text/css">
html,body{ margin: 0px; }
</style>
<!--
<script src="https://ajax.googleapis.com/ajax/libs/jquery/1.12.4/jquery.min.js"></script>
-->
<script type="text/javascript" src="http://code.jquery.com/jquery-2.2.1.min.js"></script>
<script type="text/javascript" src="http://www.openlayers.org/api/OpenLayers.js"></script>
<script type="text/javascript">
// グーローバル変数の定義
var od;
var des_lonlat;
var arr_lonlat;
</script>
<script>
function MapInit(){
map = new OpenLayers.Map("MapCanvas");
var mapnik = new OpenLayers.Layer.OSM();
map.addLayer(mapnik);
//var lonLat = new OpenLayers.LonLat(139.47552, 35.59857)
var lonLat = new OpenLayers.LonLat(139.796182, 35.654285)
.transform(
new OpenLayers.Projection("EPSG:4326"),
new OpenLayers.Projection("EPSG:900913")
);
map.setCenter(lonLat, 17);
OpenLayers.Control.Click = OpenLayers.Class(OpenLayers.Control, {
initialize: function(options) {
this.handler = new OpenLayers.Handler.Click(
this, {
'click': this.onClick
}, this.handlerOptions
);
},
onClick: function(e) {
var lonlat = map.getLonLatFromPixel(e.xy);
lonlat.transform(
new OpenLayers.Projection("EPSG:900913"),
new OpenLayers.Projection("EPSG:4326")
);
var markers = new OpenLayers.Layer.Markers("Markers");
map.addLayer(markers);
var marker = new OpenLayers.Marker(
new OpenLayers.LonLat(lonlat.lon, lonlat.lat)
.transform(
new OpenLayers.Projection("EPSG:4326"),
new OpenLayers.Projection("EPSG:900913")
)
);
markers.addMarker(marker);
$("#LonLat").html("lon:" +lonlat.lon+ " lat:" +lonlat.lat);
if (od == "arrival"){
arr_lonlat = lonlat;
alert("arr_lonlatが設定されました" + arr_lonlat.lon +" " + arr_lonlat.lat);
} else if (od == "destination"){
des_lonlat = lonlat;
alert("des_lonlatが設定されました" + des_lonlat.lon +" " + des_lonlat.lat);
}
}
});
var click = new OpenLayers.Control.Click();
map.addControl(click);
click.activate();
}
</script>
<script type="text/javascript">
$(document).ready(function () {
$("#button01").on('click', function () {
od = "destination";
alert(od + " ボタン1がクリックされました。");
});
$("#button02").on('click', function () {
od = "arrival";
alert(od + " ボタン2がクリックされました。");
});
$("#button03").on('click', function () {
od = "confirmed"
alert(od + " ボタン3がクリックされました。");
//
});
})
</script>
</head>
<body>
<div id="MapCanvas" style="width:700px;height:700px;"></div>
<div id="LonLat"></div>
<input id="button01" type="button" value="Button1"/>
<input id="button02" type="button" value="Button2" />
<input id="button03" type="button" value="Button3" />
<script type="text/javascript">MapInit();</script>
</body>
</html>